Nicola Segata e Edoardo Pasolli del Laboratorio di Metagenomica computazionale dell’Università di Trento hanno completato il più ricco catalogo di batteri e archeobatteri umani o microbioma umano mai compilato finora e l’individuazione di molte specie microbiche intestinali e orali comuni nella popolazione mondiale ma finora mai osservate, lavoro pubblicato sulla rivista scientifica “Cell”.
«Abbiamo individuato e catalogato geneticamente un grande numero di batteri e archeobatteri che costituiscono il microbioma umano, ma che finora non erano mai stati analizzati o descritti – afferma Nicola Segata -. Questo permette ora di caratterizzare una frazione sostanziale del microbioma che era rimasta finora “nascosta”. Abbiamo anche osservato molti microrganismi presenti prevalentemente in popolazioni non occidentalizzate e che in occidente sono solo raramente identificati. Probabilmente questo è una conseguenza del complesso processo di industrializzazione».
Anche i batteri, come altre forme viventi, evolvono e vengono selezionati con il cambiare dell’ambiente, dell’alimentazione e dello stile di vita e in alcuni casi rischiano di estinguersi. Il team di ricerca si è messo sulle tracce di alcuni di essi. Lo studio è un intreccio di genomica, microbiologia e big data. Per oltre due anni ha coinvolto ricercatori e ricercatrici del Dipartimento Cibio dell’Università di Trento assieme a studenti e studentesse della laurea magistrale in Quantitative and Computational Biology dell’Ateneo. Ed è frutto di una collaborazione internazionale nella quale hanno avuto un ruolo rilevante docenti di Harvard che studiano popolazioni non occidentalizzate del Madagascar.
Segata nelle sue ricerche si occupa del microbioma umano. «Si tratta di quell’insieme di batteri, virus, funghi e parassiti che si trovano in particolare nell’intestino, nella bocca, sulla pelle e nell’apparato genitale. Il microbioma, con cui le cellule umane vivono in simbiosi, svolge funzioni cruciali per il corpo, dal metabolismo all’attività sull’asse intestino-cervello, dalla protezione diretta contro organismi patogeni alla regolazione del sistema immunitario. Si è dimostrato che il microbioma ha un ruolo nell’insorgenza di alcuni tumori e nel successodella immunoterapia contro il cancro».
L’approccio suo e del suo gruppo di ricerca allo studio del microbioma si chiama metagenomica computazionale: studiano il microbioma analizzando il suo contenuto genetico. Da una goccia di saliva, tampone cutaneo o grammo di feci estraggono il DNA di tutti i microrganismi presenti, lo sequenziano con le macchine ad alta precisione di cui è dotata l’Università di Trento. Con speciali super-computer analizzano poi il DNA sequenziato per ricostruire composizione e dinamiche del microbioma.
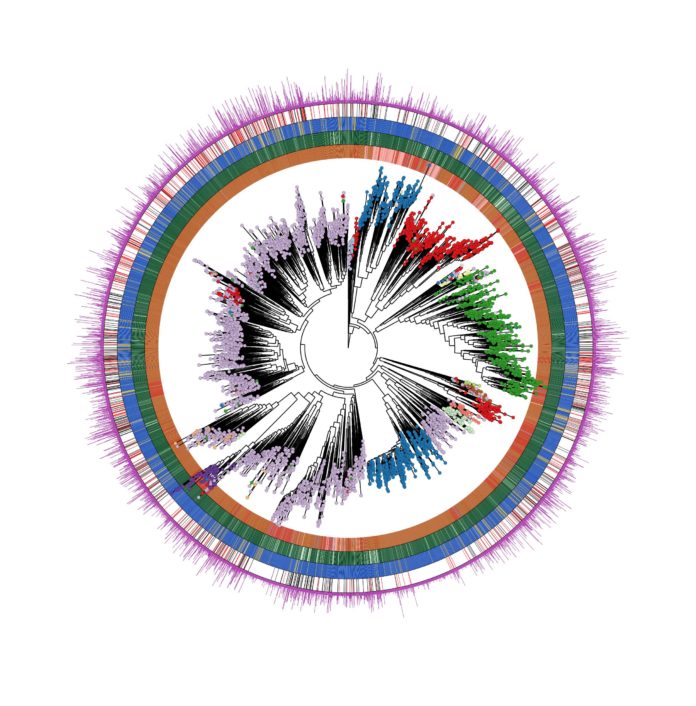
Segata entra nel dettaglio della ricerca: «con questo studio che è frutto del lavoro di tutto il team multidisciplinare a CIBIO che comprende microbiologi, statistici, e informatici, abbiamo individuato quasi 5.000 specie che catalogano gli oltre 154.000 genomi ricostruiti e descrivono il microbioma umano al variare di età, distretto corporeo, dieta, malattia. Ogni individuo possiede fino a diverse centinaia di queste specie. Una grossa frazione di queste 5.000 specie (il 77%) erano precedentemente sconosciute. Alcune di queste specie sono molto prevalenti nella popolazione e la loro scoperta è la base di partenza per poter testare il loro ruolo in malattie autoimmuni, gastro-intestinali, e oncologiche. Per arrivare a questi risultati abbiamo analizzato una mole di dati estremamente grande e diversificata per provenienza geografica, stile di vita, età. In tutto abbiamo analizzato 9.428 campioni da tutti i continenti».

E sulla scoperta e analisi di uno di questi batteri precisa: «la specie sconosciuta più comune, che abbiamo chiamato “Cibiobacter qucibialis”, è il settimo organismo intestinale più prevalente nella popolazione e l’abbiamo individuata ricostruendo più di 1.800 genomi. Questa specie potrà essere di grande importanza per la comprensione delle funzioni del microbioma umano e come sfruttarlo in biomedicina».
Infine un approfondimento sulle differenze tra popolazioni: «ci siamo soffermati sulle popolazioni non occidentalizzate che non hanno cioè accesso alle diete ad alto contenuto di grassi, agli antibiotici e altri medicinali e sottoposte a condizioni igieniche differenti. Molti nuovi microrganismi scoperti nelle popolazioni non occidentalizzate tendono a non essere più identificabili nelle nostre popolazioni occidentali. Il lavoro ha quindi posto i presupposti per studiare queste specie e capire se possano essere legate all’aumento di malattie autoimmuni, allergie e malattie complesse del mondo occidentale. Pensiamo anche che sia necessario cercare di coltivare e conservare queste specie che, in futuro, si potrebbe pensare di provare a reintrodurre nelle popolazioni occidentalizzate».
Per rimanere sempre aggiornato con le ultime notizie de “Il NordEst Quotidiano”, iscriviti al canale Telegram
o vai su Twitter
@nestquotidiano
https://twitter.com/nestquotidiano
© Riproduzione Riservata